Appendix D
## [1] "2025-11-05"A.2 COVID 19 Cumulativo
Usa los datos de COVID-19 en Puerto Rico (PR) del Departamento de Salud de PR.
## # A tibble: 6 × 18
## ...1 Fecha Muertes IncrementoMuertes CasosPCR_Salud IncCasosPCR_Salud
## <dbl> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 1 3/12/20 0 0 NA NA
## 2 2 3/13/20 0 0 NA NA
## 3 3 3/14/20 0 0 NA NA
## 4 4 3/15/20 0 0 NA NA
## 5 5 3/16/20 0 0 NA NA
## 6 6 3/17/20 0 0 NA NA
## # ℹ 12 more variables: CasosSaludNuevo <dbl>, IncCasosSaludNuevo <dbl>,
## # HospitCOV19 <dbl>, CamasICU <dbl>, CamasICU_disp <dbl>, Ventiladores <dbl>,
## # MuertesSalud <dbl>, IncMueSalud <dbl>, VacDoses <dbl>, VacAdm <dbl>,
## # N1MoreDoses <dbl>, N2Doses <dbl>- Calcula los promedios en periodos de 7 (dias; promedio corriendo) y añádele a tu dataframe
_ Usa la columna de IncCasosSaludNuevo para todos los cálculos
- Enseñe las primeras 20 filas de tu dataframe con las nuevas columnas añadidas
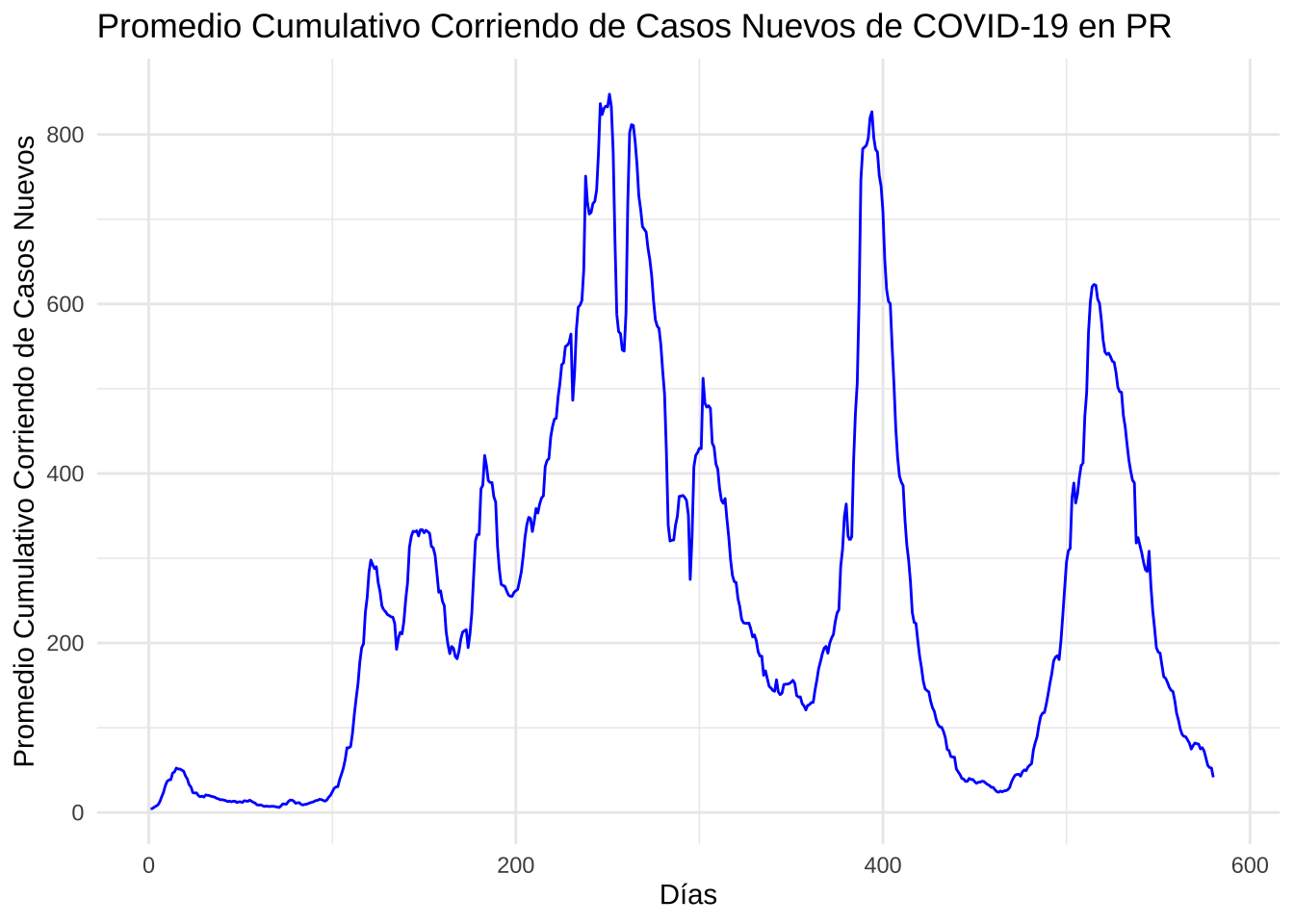
- Haz una figura que muestra el cambio en el promedio cumulativo corriendo de casos nuevos.