Capítulo16 Importar datos
Fecha de la ultima revisión
## [1] "2025-11-05"16.1 Datos importado de la web.
16.1.1 Usar datos que provienen de una base de datos del web.
Aquí un website que recopila la cantidad de personas vacunados en los EEUU.
Seleccionar solamente los datos de Puerto Rico
16.1.2 COVID world
Otro website de datos de COVID, estos representa los datos de todos los paises del mundo
Para encontrar datos de PR vea el siguiente enlace: Este sitio del gobierno de Puerto Rico almacen datos muchos diferentes tipos que estan relacionado a la isla.
https://datos.estadisticas.pr/dataset?res_format=CSV
Nota que para leer los datos de un archivo CSV, se usa la función “fread()” del paquete “data.table”.
- Cuando entre a la pagina, selecciona uno de los archivos, por ejemplo Facultad -Conservatorio de Musica
- Presione el boton “Explore” y selecciona “Preview”
- Arriba de la pagina vera el enlace de https://……… para poner en un script.
- Copie y pegue en el script.
Otra alternativa es bajar los datos en formato .csv o Excel y subirlo a su proyecto en RStudio.
16.2 Ejercicio
- Selecciona solamente los datos de los bebes que nacieron primer mes del año, pero agrupado por el día de la semana y contabilizado
COVID_PR
- Calcula la cantidad de muertes por día en PR, y el máximo de muerto en un día por COVID
16.3 Datos importando de su computadora
16.3.1 Temas: Funciones para importar datos con el paquete readr
- read_csv() # sepeardo por coma
- read_csv2() # separado por punto y coma
- read_tsv() # separado por tab
- read_delim() # separado por delimitador
- read_fwf()
- read_width()
- read_positions()
- read_table()
library(tidyverse)
library(readr)
Newborn_Karn_Penrose <- read_csv("Datos/Newborn_Karn_Penrose.csv")
#library(readxl)
#Newborn_Karn_Penrose1 <- read_excel("~/Google Drive/GitHub_Google_Drive/GitHub/Vintage_DATA/DATA/Human_Birth_Weight/Newborn_Karn_Penrose.xlsx", sheet = 3)
head(Newborn_Karn_Penrose)## # A tibble: 6 × 4
## Mother_age Count_Non_survivors Count_Total_Birth Birth_weight
## <dbl> <dbl> <dbl> <dbl>
## 1 16 0 1 9.5
## 2 16 0 2 9
## 3 16 0 1 8.5
## 4 16 0 3 8
## 5 16 0 7 7.5
## 6 16 0 2 7Hacer una columna de la proporción de los niños que no sobrevivieron.
Newborn_Karn_Penrose %>% select(Count_Non_survivors, Count_Total_Birth) %>% arrange(Count_Total_Birth, Count_Non_survivors) %>% mutate(Perc_sobrevivieron = Count_Non_survivors/Count_Total_Birth)
## # A tibble: 261 × 5
## Mother_age Count_Non_survivors Count_Total_Birth Birth_weight Percent_Decease
## <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 16 0 1 9.5 0
## 2 16 0 2 9 0
## 3 16 0 1 8.5 0
## 4 16 0 3 8 0
## 5 16 0 7 7.5 0
## 6 16 0 2 7 0
## 7 16 0 4 6.5 0
## 8 16 0 2 6 0
## 9 16 0 2 5.5 0
## 10 16 0 1 5 0
## # ℹ 251 more rowsNewborn_Karn_Penrose %>% select(Count_Non_survivors, Count_Total_Birth, Birth_weight) %>% arrange(Count_Total_Birth, Count_Non_survivors) %>% mutate(porc_sobrevivieron = Count_Non_survivors / Count_Total_Birth)%>% ggplot(aes(x= Birth_weight, y=porc_sobrevivieron))+ geom_point()+ geom_smooth()
Newborn_Karn_Penrose %>%
mutate(Percent_Decease = Count_Non_survivors/Count_Total_Birth) %>%
ggplot(aes(Birth_weight, Percent_Decease)) +
geom_point() +
geom_smooth()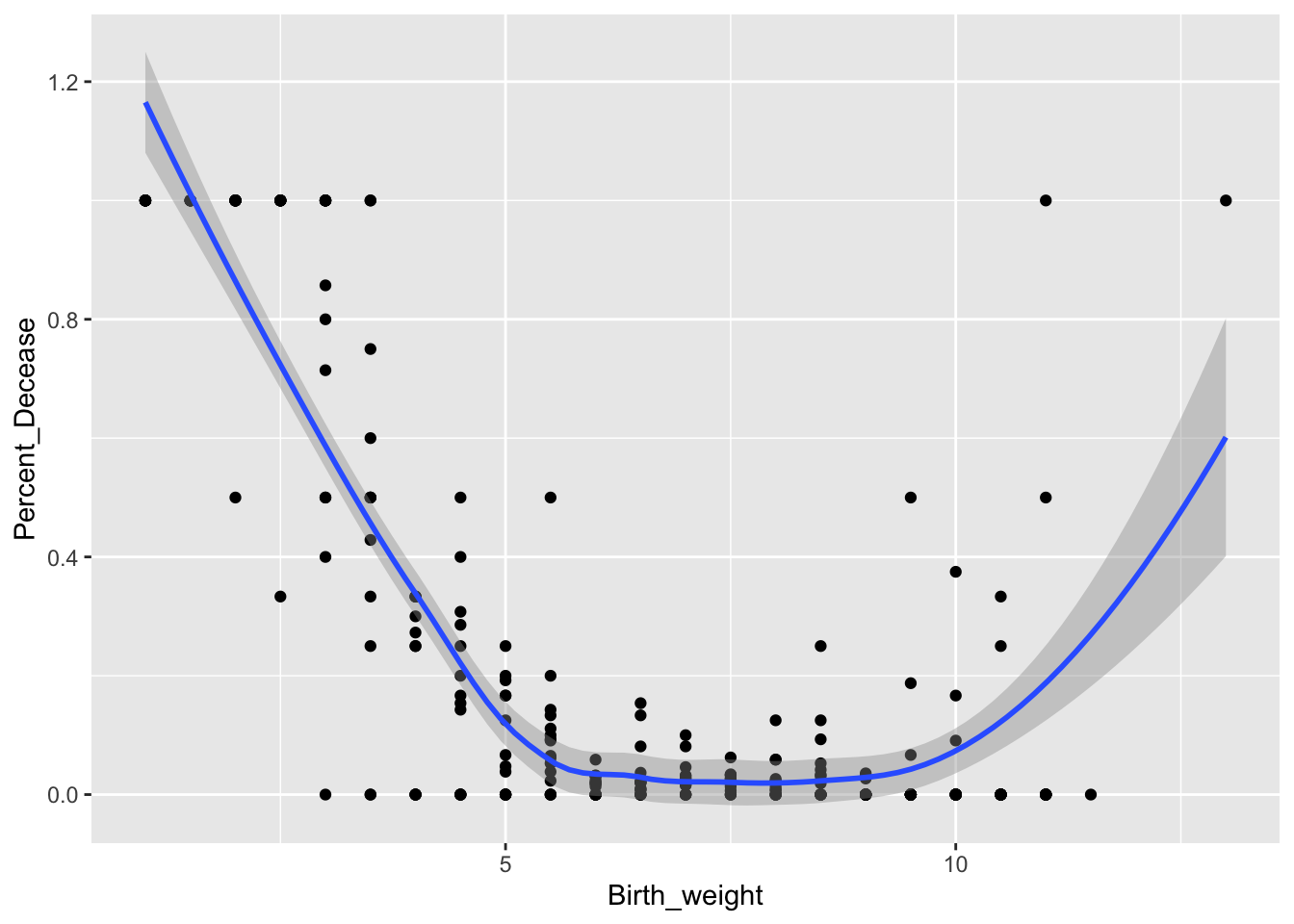
Newborn_Karn_Penrose %>%
mutate(Percent_Decease = Count_Non_survivors/Count_Total_Birth) %>%
ggplot(aes(Birth_weight, Percent_Decease)) +
geom_point() +
geom_smooth()+
facet_wrap(~Mother_age)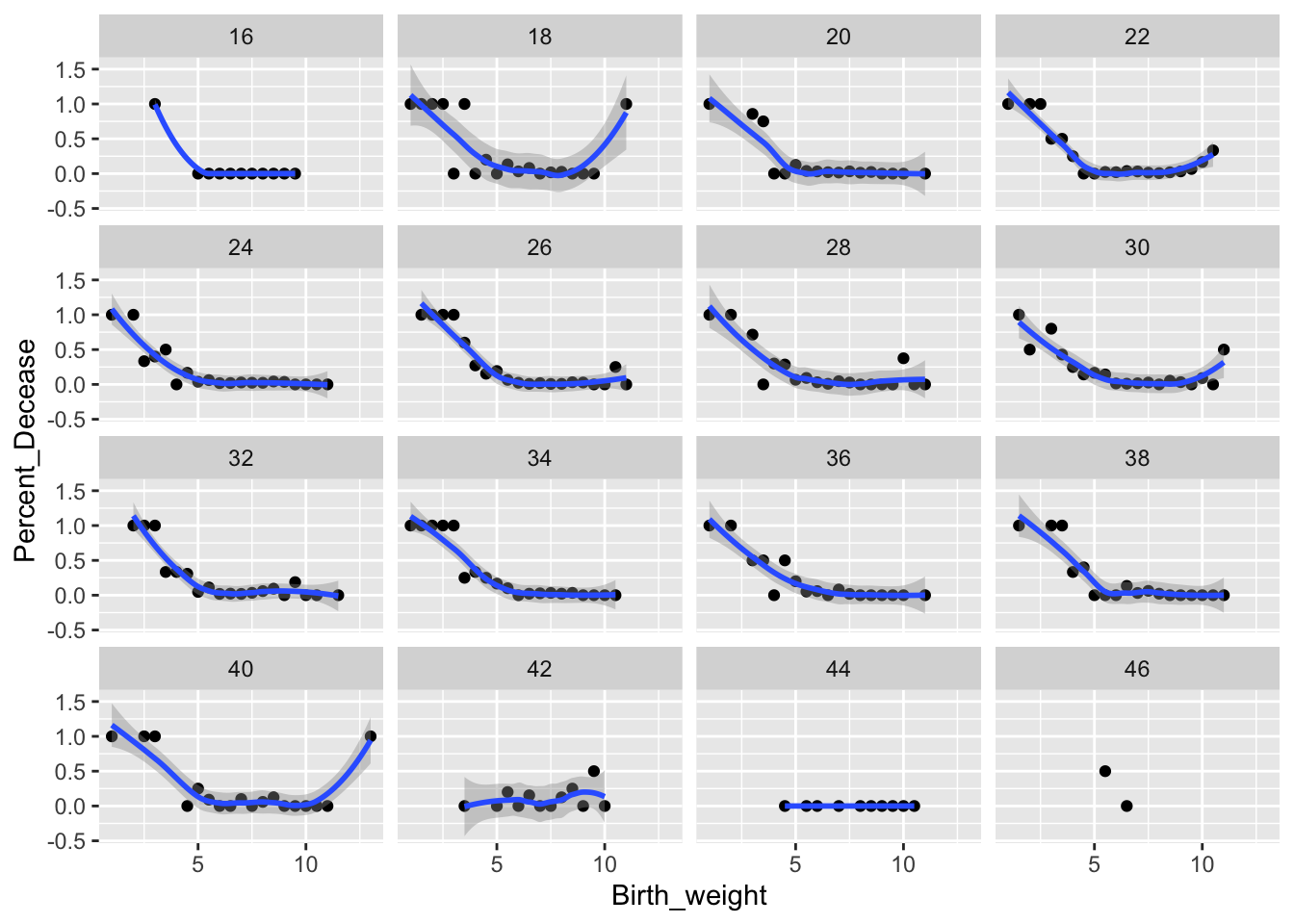
Newborn_Karn_Penrose %>%
mutate(Percent_Decease = Count_Non_survivors/Count_Total_Birth) %>%
ggplot(aes(Birth_weight, Percent_Decease, color=Mother_age)) +
geom_point(aes(color=factor(Mother_age))) +
geom_smooth(se=FALSE, aes(color=factor(Mother_age)))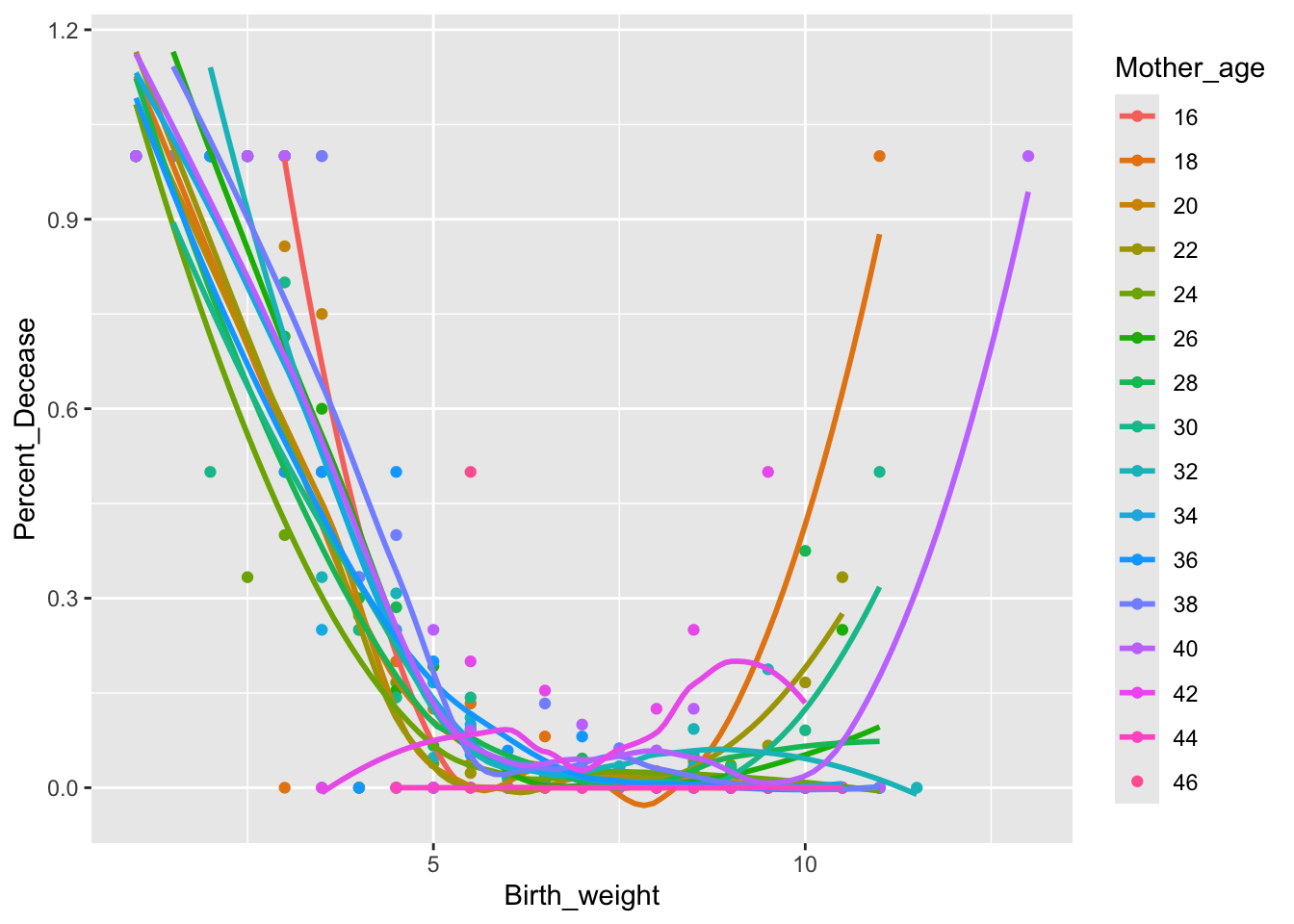
16.5 Segmentar un vector
- str(parse_logical())
## [1] "TRUE" "FALSE" "NA"## logi [1:3] TRUE FALSE NA## [1] "Carlos" "Kelvin"## logi [1:2] NA NA
## - attr(*, "problems")= tibble [2 × 4] (S3: tbl_df/tbl/data.frame)
## ..$ row : int [1:2] 1 2
## ..$ col : int [1:2] NA NA
## ..$ expected: chr [1:2] "1/0/T/F/TRUE/FALSE" "1/0/T/F/TRUE/FALSE"
## ..$ actual : chr [1:2] "Carlos" "Kelvin"## logi [1:3] TRUE FALSE NA## [1] "1" "2" "3"## [1] NA## int [1:3] 1 2 3## int [1:3] 1 2 3## Date[1:2], format: "2010-01-01" "1979-10-14"## [1] 4000 500## [1] 100## [1] 20## [1] 123.45- str(parse_integer())
- str(parse_date())
16.7 Caderna de texto
parse_character()
charToRaw()
## [1] 52 61 6d 6ex1 <- "El Ni\xf1o was particularly bad this year"
x2 <- "\x82\xb1\x82\xf1\x82\xc9\x82\xbf\x82\xcd"
x3 <- "The boy was particularly bad this year"
library(readr)
parse_character(x1, locale = locale(encoding = "Latin1"))## [1] "El Niño was particularly bad this year"#> [1] "El Niño was particularly bad this year"
parse_character(x2, locale = locale(encoding = "Shift-JIS"))## [1] "こんにちは"## [1] "TRUE" "FALSE" NA- guess_encoding(charToRaw())
## # A tibble: 1 × 2
## encoding confidence
## <chr> <dbl>
## 1 ASCII 1